Funções e módulos¶
Introdução¶
No programa seguinte, dada uma sequência de DNA pretende-se mostrar a sequência, a sequência complementar e o complemento reverso na forma de codões separados por um hífen.
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 | seq = "AGCTGGATCCTGAACGCATAGACTAGCATGGGACTAAAGGTCCATTACTGA" trans = {'A':'T', 'T':'A', 'C':'G', 'G':'C'} comp = ''.join([trans[b] for b in seq]) comp_rev = ''.join(reversed(comp)) seq_cods = [seq[i:i+3] for i in range(0, len(seq), 3)] chain_seq = '-'.join(seq_cods) print("5'-" + chain_seq +"-3'") comp_cods = [comp[i:i+3] for i in range(0, len(comp), 3)] chain_comp = '-'.join(comp_cods) print("3'-" + chain_comp + "-5'") comp_rev_cods = [comp_rev[i:i+3] for i in range(0, len(comp_rev), 3)] chain_comp_rev = '-'.join(comp_rev_cods) print('Complemento reverso') print("5'-" + chain_comp_rev + "-3'") |
5'-AGC-TGG-ATC-CTG-AAC-GCA-TAG-ACT-AGC-ATG-GGA-CTA-AAG-GTC-CAT-TAC-TGA-3' 3'-TCG-ACC-TAG-GAC-TTG-CGT-ATC-TGA-TCG-TAC-CCT-GAT-TTC-CAG-GTA-ATG-ACT-5' Complemento reverso 5'-TCA-GTA-ATG-GAC-CTT-TAG-TCC-CAT-GCT-AGT-CTA-TGC-GTT-CAG-GAT-CCA-GCT-3'
A parte do programa nas linhas 7 e 8
seq_cods = [seq[i:i+3] for i in range(0, len(seq), 3)] chain_seq = '-'.join(seq_cods)
repete-se de uma forma análoga para a sequência complementar e para o
complemento reverso nas linhas 11 e 12 e também nas linhas 15 e 16. O que
muda é a sequência sobre a qual estão a ser aplicadas (seq nas linhas 7 e 8,
comp nas linhas 11 e 12 e comp_rev nas linhas 15 e 16).
O objetivo de cada grupo destas duas linhas é muito claro: separar uma sequência (string) numa listas de codões e depois construír uma string com os codões separados por um hífen.
Como podemos não repetir o "texto" destas três partes do programa, que são muito semelhantes?
Funções¶
As funções (também conhecidas como subprogramas, subrotinas, isto é, mini-programas dentro de programas) são a solução para estes casos.
Já vimos várias funções, sempre disponíveis ou disponíveis após importação de módulos:
a = 'Uma pequena string' n = len(a) f = int(4.2) nA = a.count('A') a = [] a.append(33) import math l = math.log(2.0)
Definição de funções (def)¶
Podemos escrever outras funções para "aumentar" a linguagem.
Tal como na matemática, as funções transformam objetos noutros objetos:
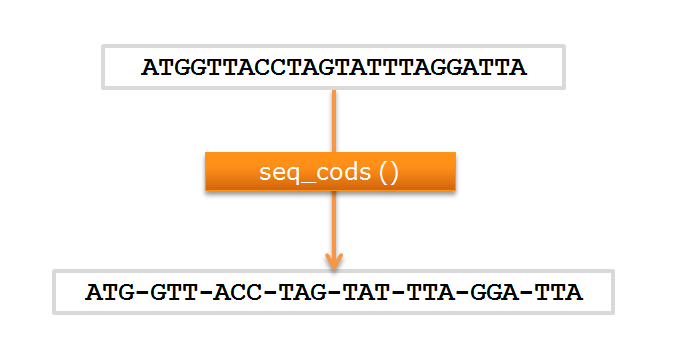
Mas, tal como na matemática, as funções são escritas para atuar sobre
objetos genéricos (x):

Problema: escrever uma função que, dada uma sequência, devolva a
sequência com os codões separados por -.
def seqcods(x): cods = [x[i:i+3] for i in range(0,len(x),3)] comhifen = '-'.join(cods) return comhifen
Anatomia de uma função¶

A definição de uma função (def) não executa nada imediatamente.
É necessário chamar (ou "invocar") a função para esta ser usada:
def seqcods(x): cods = [x[i:i+3] for i in range(0,len(x),3)] comhifen = '-'.join(cods) return comhifen a = "ATGGTTACCTAGTATTTAGGATTA" print(a) # A função é chamada aqui: s = seqcods(a) print(s)
ATGGTTACCTAGTATTTAGGATTA ATG-GTT-ACC-TAG-TAT-TTA-GGA-TTA
Nota
O comando return pode "devolver" uma expressão complicada
(não só o nome de um objeto)
def seqcods(x): return '-'.join( [x[i:i+3] for i in range(0,len(x),3)]) a = "ATGGTTACCTAGTATTTAGGATTA" print(a) # A função é chamada aqui: s = seqcods(a) print(s)
ATGGTTACCTAGTATTTAGGATTA ATG-GTT-ACC-TAG-TAT-TTA-GGA-TTA
Em resumo:
A linha
def seqcods(x):
"regista" uma nova função, chamada seqcods, que pode ser usada em
qualquer ponto do programa, da forma seguinte:
s = seqcods(a)
Entrada e saída de valores quando uma função é chamada:

Exemplo: função factorial():
def factorial(n): res = 1 for k in range(2,n+1): res = res * k return res print(factorial(200))
788657867364790503552363213932185062295135977687173263294742533244359449963403342920304284011984623904177212138919638830257642790242637105061926624952829931113462857270763317237396988943922445621451664240254033291864131227428294853277524242407573903240321257405579568660226031904170324062351700858796178922222789623703897374720000000000000000000000000000000000000000000000000
Vários tipos de funções¶
a = 'Uma pequena string' #1 argumento, 1 resultado print( len(a) ) #1 arg, 1 res, associada a um objeto (string a) print( a.count('a') ) #0 arg, 1 res, associada a um objeto (string a) print( a.upper() )
18 2 UMA PEQUENA STRING
#1 arg, 0 res, associada a um objeto (lista b) # modifica o objeto (a lista b) b = [12, 24] print( b.append(36) ) print(b)
None [12, 24, 36]
Além da função .append(), recordar que as listas têm outras duas
funções deste tipo, que modificam a lista sem produzir nenhum resultado
(o resultado é a constante None): .reverse() e .sort().
b = [12, 24, 36] print(b) b.reverse() print(b) b.sort() print(b)
[12, 24, 36] [36, 24, 12] [12, 24, 36]
As funções podem ter mais de um argumento.
O resultado pode não ser apenas um número ou uma string: as funções podem devolver uma lista inteira, um dicionário ou outros objetos mais complexos.
import math print( math.log(64, 2) ) import time x = time.localtime(time.time()) print(x)
6.0 time.struct_time(tm_year=2018, tm_mon=4, tm_mday=8, tm_hour=18, tm_min=39, tm_sec=43, tm_wday=6, tm_yday=98, tm_isdst=1)
Problema: eliminar valores de uma lista que pertençam a uma "lista negra"
def elimin_black(uma_lista, black_list): res = [i for i in uma_lista if i not in black_list] return res a = [1, 2, 4, 'um', 'dois', 3, 42, 'quatro'] print(a) black = [1, 2, 'um', 'dois'] print ('\nA eliminar:', black) clean = elimin_black(a, black) print(clean)
[1, 2, 4, 'um', 'dois', 3, 42, 'quatro'] A eliminar: [1, 2, 'um', 'dois'] [4, 3, 42, 'quatro']
Problema: dado um nome de um ficheiro de texto, escrever uma
função para ler o conteúdo do ficheiro para uma lista de linhas sem o
\n no final, excluíndo as linhas vazias.
def ler_fich(nome): linhas = [] with open(nome) as a: for linha in a: linha = linha.strip() if len(linha) > 0: linhas.append(linha) return linhas todos = ler_fich('gre3.txt') for i in todos: print(i)
>sp|P38715|GRE3_YEAST NADPH-dependent aldose reductase GRE3 OS=Saccharomyces cerevisiae (strain ATCC 204508 / S288c) GN=GRE3 PE=1 SV=1 MSSLVTLNNGLKMPLVGLGCWKIDKKVCANQIYEAIKLGYRLFDGACDYGNEKEVGEGIR KAISEGLVSRKDIFVVSKLWNNFHHPDHVKLALKKTLSDMGLDYLDLYYIHFPIAFKYVP FEEKYPPGFYTGADDEKKGHITEAHVPIIDTYRALEECVDEGLIKSIGVSNFQGSLIQDL LRGCRIKPVALQIEHHPYLTQEHLVEFCKLHDIQVVAYSSFGPQSFIEMDLQLAKTTPTL FENDVIKKVSQNHPGSTTSQVLLRWATQRGIAVIPKSSKKERLLGNLEIEKKFTLTEQEL KDISALNANIRFNDPWTWLDGKFPTFA
Problema: eliminar valores repetidos numa lista
def elimin_reps(uma_lista): res = [] for i in uma_lista: if i not in res: res.append(i) return res uma_lista = [1, 2, 4, 7, 7, 5, 8, 8, 9, 10] print(uma_lista) clean = elimin_reps(uma_lista) print(clean)
[1, 2, 4, 7, 7, 5, 8, 8, 9, 10] [1, 2, 4, 7, 5, 8, 9, 10]
Note-se que na função é criada uma lista nova:
res = []
...
res.append(i)
e é esta lista que é o resultado da função.
Problema: eliminar valores repetidos numa lista, mas sem ser
devolvida uma lista nova como resultado. Isto é, a função recebe uma
lista e modifica-a, não havendo return.
def elimin_reps2(uma_lista): res = [] for i in uma_lista: if i not in res: res.append(i) uma_lista[:] = res uma_lista = [1, 2, 4, 7, 7, 5, 8, 8, 9, 10] print('Antes', uma_lista) elimin_reps2(uma_lista) # não havendo return NÃO se dá um nome # ao resultado print('Depois', uma_lista)
Antes [1, 2, 4, 7, 7, 5, 8, 8, 9, 10] Depois [1, 2, 4, 7, 5, 8, 9, 10]
O que significa uma_lista[:] = res ?
Usa-se um slice para toda a lista (uma_lista[:] significa todos os
elementos do princípio o fim)e atribuí-se a esse slice uma lista nova.
Assim, toda a lista é modificada.
Nota: não é possível usar esta técnica com strings. As strings são imutáveis.
Se as funções tiverem resultados é possível usá-las em cadeia:
def elimin_reps(uma_lista): res = [] for i in uma_lista: if i not in res: res.append(i) return res def elimin_black(uma_lista, black_list): return [i for i in uma_lista if i not in black_list] a = [1, 2, 4, 'um', 'dois', 3, 3, 37, 42, 42, 'quatro'] black = [1, 2, 'um', 'dois'] clean = elimin_reps(elimin_black(a, black)) print(clean)
[4, 3, 37, 42, 'quatro']
Âmbito dos nomes¶
def recta(m, b, x): print('Para x =', x) print('com m =', m) print('com b =', b) r1 = m*x r0 = b return(r1 + r0) x, c1, c0 = 2.0, 3.0, 2.0 res = recta(c1, c0, x) print('Resultado:', res)
Para x = 2.0 com m = 3.0 com b = 2.0 Resultado: 8.0
Este programa corre sem problemas.
Note-se que podemos usar a função print() dentro de uma função.
def recta(m, b, x): r1, r0 = m*x, b return r1 + r0 m, b, x = 2.0, 3.0, 2.0 res = recta(m, b, x) print('Para x =', x, 'm =', m, 'b =', b) print('m*x =', r1, 'b =', r0) print('Resultado:', res)
Para x = 2.0 m = 2.0 b = 3.0
:
---------------------------------------------------------------------------
NameError Traceback (most recent call last)
<ipython-input-17-f26c93717bbe> in <module>()
7
8 print('Para x =', x, 'm =', m, 'b =', b)
----> 9 print('m*x =', r1, 'b =', r0)
10 print('Resultado:', res)
NameError: name 'r1' is not defined
O que se passou aqui?
Os nomes usados dentro da função r1 e r0 são locais: pertencem ao
âmbito da função.
Qualquer parte do programa "exterior" à função não consegue "ver" esses nomes. Daí o erro durante a execução.
O mesmo acontece aos próprios nomes locais dos argumentos da função:
def recta2(m2, b2, x): r1, r0 = m2*x, b2 return r1 + r0 m, b, x = 2.0, 3.0, 2.0 res = recta2(m, b, x) print('Para x =', x, 'm2 =', m2, 'b2 =', b2) print('Resultado:', res)
:
---------------------------------------------------------------------------
NameError Traceback (most recent call last)
<ipython-input-18-92c7134da27b> in <module>()
6 res = recta2(m, b, x)
7
----> 8 print('Para x =', x, 'm2 =', m2, 'b2 =', b2)
9 print('Resultado:', res)
NameError: name 'm2' is not defined
def recta(m, b, x): print('Dentro da função --------') print('m =', m, 'b =', b, 'x =', x) print('-------------------------') x = m * x + b return x m = 2 b = 2 x = 4 res = recta(m + 3, b + 3, x * x) print('m =', m, 'b =', b, 'x =', x) print('\nResultado:', res)
Dentro da função -------- m = 5 b = 5 x = 16 ------------------------- m = 2 b = 2 x = 4 Resultado: 85
Este programa corre sem problemas!
Mas cada um dos nomes m, b, x é usado em dois contextos e tem
valores diferentes:
- O contexto local, quando estão "dentro" da função.
- O contexto global, quando estão "fora da função".
Fora da função, os valores globais são:
m = 2 b = 2 x = 4
Estes valores não são modificados fora da função e são apresentados pela
função print() no final.
Dentro da função estes nomes são, em primeiro lugar, usados como os argumentos da função.
Pela maneira como a função é chamada, estes valores são:
m = 5 b = 5 x = 16
O nome x é modificado dentro da função (x = m * x + b) ficando com o
valor final 85 e é este valor que é o resultado da função (return x).
Quando a função termina e estamos de novo "de fora" da função, o valor
de x volta a ser 4, uma vez que voltamos a um contexto "global".
Argumentos de tipo _palavra-chave¶
def mix(a=1, b=0): c = a + b print('a =', a, 'b =', b, '--> return =', c) return c x = mix() x = mix(b=3) x = mix(a=2, b=3) x = mix(2,3)
a = 1 b = 0 --> return = 1 a = 1 b = 3 --> return = 4 a = 2 b = 3 --> return = 5 a = 2 b = 3 --> return = 5
def factorial(n, trace=False): p = 1 for i in range(2,n+1): p = p * i if trace: print(i, p) return p f20 = factorial(20) print('O factorial de 20 é', f20)
O factorial de 20 é 2432902008176640000
def factorial(n, trace=False): p = 1 for i in range(2,n+1): p = p * i if trace: print(i, p) return p f20 = factorial(20, trace=True) print('O factorial de 20 é', f20)
2 2 3 6 4 24 5 120 6 720 7 5040 8 40320 9 362880 10 3628800 11 39916800 12 479001600 13 6227020800 14 87178291200 15 1307674368000 16 20922789888000 17 355687428096000 18 6402373705728000 19 121645100408832000 20 2432902008176640000 O factorial de 20 é 2432902008176640000
Módulos e import¶
Como vimos, muitas funções estão imediatamente disponíveis quando começamos a escrever um programa.
Estas funções podem ser "livres" (por exemplo a função len()) ou
associadas a objetos (por exemplo, s.split() que está associada à
string s).
A linguagem Python permite a organização dos programas em módulos.
Módulos são ficheiros .py (ou ficheiros pré-compilados) que contêm coleções de funções úteis e relacionadas entre si, podendo também conter alguns outros objetos que não são funções, mas que contêm informação de suporte.
Os módulos são também essenciais no aumento da funcionalidade da própria linguagem python: com a distribuição oficial do Python e em algumas distribuições de "Python científico" são tipicamente disponibilizados centenas de módulos, em que cada módulo reúne a funcionalidade adicional relacionada com um propósito particular.
Por exemplo, o módulo math reúne funções associadas a cálculos
matemáticos (e as constantes \pi e e).
O módulo time reúne funções e constantes relacionadas com a utilização
da data e hora.
A documentação sobre a coleção de módulos que são são disponibilizados em qualquer distribuição da linguagem Python (a chamada Biblioteca padrão (em inglês Standard Library) pode ser encontrada na documentação oficial
Por outro lado, quem escreve módulos novos pode submetê-los ao Python Package Index, conhecido como PyPi. Trata-se de um imenso depósito de módulos com contribuições de milhares de autores e em permanente crescimento.
Exemplo da construção de um módulo¶
def readFASTA(filename): """This function reads a FASTA format file and returns a pair of strings with the header and the sequence """ with open(filename) as a: lines = [line.strip() for line in a] lines = [line for line in lines if len(line) > 0] if lines[0].startswith('>'): return lines[0], ''.join(lines[1:]) else: return '', ''.join(lines) h, s = readFASTA("gre3.txt") print(f'Header:\n{h}\n\nSequence:\n{s}')
Header: >sp|P38715|GRE3_YEAST NADPH-dependent aldose reductase GRE3 OS=Saccharomyces cerevisiae (strain ATCC 204508 / S288c) GN=GRE3 PE=1 SV=1 Sequence: MSSLVTLNNGLKMPLVGLGCWKIDKKVCANQIYEAIKLGYRLFDGACDYGNEKEVGEGIRKAISEGLVSRKDIFVVSKLWNNFHHPDHVKLALKKTLSDMGLDYLDLYYIHFPIAFKYVPFEEKYPPGFYTGADDEKKGHITEAHVPIIDTYRALEECVDEGLIKSIGVSNFQGSLIQDLLRGCRIKPVALQIEHHPYLTQEHLVEFCKLHDIQVVAYSSFGPQSFIEMDLQLAKTTPTLFENDVIKKVSQNHPGSTTSQVLLRWATQRGIAVIPKSSKKERLLGNLEIEKKFTLTEQELKDISALNANIRFNDPWTWLDGKFPTFA
Vamos supor que esta seria a primeira de uma coleção de funções relacionadas com o processamento de ficheiros de texto contendo sequências usados em bioinformática (uma sequência em FASTA é apenas um exemplo).
Podemos criar um ficheiro biosequences.py contendo essa função:
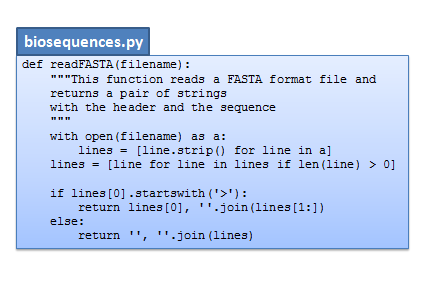
Este ficheiro constitui um módulo que pode ser usado num programa.
Para isso, é necessário usar o comando import:
import biosequences h, s = biosequences.readFASTA("gre3.txt") print(f'Header:\n{h}\n\nSequence:\n{s}')
Header: >sp|P38715|GRE3_YEAST NADPH-dependent aldose reductase GRE3 OS=Saccharomyces cerevisiae (strain ATCC 204508 / S288c) GN=GRE3 PE=1 SV=1 Sequence: MSSLVTLNNGLKMPLVGLGCWKIDKKVCANQIYEAIKLGYRLFDGACDYGNEKEVGEGIRKAISEGLVSRKDIFVVSKLWNNFHHPDHVKLALKKTLSDMGLDYLDLYYIHFPIAFKYVPFEEKYPPGFYTGADDEKKGHITEAHVPIIDTYRALEECVDEGLIKSIGVSNFQGSLIQDLLRGCRIKPVALQIEHHPYLTQEHLVEFCKLHDIQVVAYSSFGPQSFIEMDLQLAKTTPTLFENDVIKKVSQNHPGSTTSQVLLRWATQRGIAVIPKSSKKERLLGNLEIEKKFTLTEQELKDISALNANIRFNDPWTWLDGKFPTFA
Há mais duas maneiras de utilizar o comando import:
from biosequences import readFASTA h, s = readFASTA("gre3.txt") print(f'Header:\n{h}\n\nSequence:\n{s}')
Header: >sp|P38715|GRE3_YEAST NADPH-dependent aldose reductase GRE3 OS=Saccharomyces cerevisiae (strain ATCC 204508 / S288c) GN=GRE3 PE=1 SV=1 Sequence: MSSLVTLNNGLKMPLVGLGCWKIDKKVCANQIYEAIKLGYRLFDGACDYGNEKEVGEGIRKAISEGLVSRKDIFVVSKLWNNFHHPDHVKLALKKTLSDMGLDYLDLYYIHFPIAFKYVPFEEKYPPGFYTGADDEKKGHITEAHVPIIDTYRALEECVDEGLIKSIGVSNFQGSLIQDLLRGCRIKPVALQIEHHPYLTQEHLVEFCKLHDIQVVAYSSFGPQSFIEMDLQLAKTTPTLFENDVIKKVSQNHPGSTTSQVLLRWATQRGIAVIPKSSKKERLLGNLEIEKKFTLTEQELKDISALNANIRFNDPWTWLDGKFPTFA
from biosequences import * h, s = readFASTA("gre3.txt") print(f'Header:\n{h}\n\nSequence:\n{s}')
Header: >sp|P38715|GRE3_YEAST NADPH-dependent aldose reductase GRE3 OS=Saccharomyces cerevisiae (strain ATCC 204508 / S288c) GN=GRE3 PE=1 SV=1 Sequence: MSSLVTLNNGLKMPLVGLGCWKIDKKVCANQIYEAIKLGYRLFDGACDYGNEKEVGEGIRKAISEGLVSRKDIFVVSKLWNNFHHPDHVKLALKKTLSDMGLDYLDLYYIHFPIAFKYVPFEEKYPPGFYTGADDEKKGHITEAHVPIIDTYRALEECVDEGLIKSIGVSNFQGSLIQDLLRGCRIKPVALQIEHHPYLTQEHLVEFCKLHDIQVVAYSSFGPQSFIEMDLQLAKTTPTLFENDVIKKVSQNHPGSTTSQVLLRWATQRGIAVIPKSSKKERLLGNLEIEKKFTLTEQELKDISALNANIRFNDPWTWLDGKFPTFA
Esta última maneira é desaconselhada: um módulo pode conter centenas de funções e, por isso, podem ser importados centenas de novos nomes para um programa, que podem entrar em conflito com outros nomes iguais utilizados no programa.
Um módulo pode conter outros objetos para além de funções.
A pensar em algumas operações que poderiam ser realizadas com sequências biológicas, o ficheiro biosequences.py poderia ser ampliado com as seguintes atribuições:
.....
basesDNA = 'ATGC'
basesRNA = 'AUGC'
aa_residues = "ACDEFGHIKLMNPQRSTVWY"
complement = { 'A':'T', 'T':'A', 'G':'C', 'C':'G'}
complementRNA = { 'A':'U', 'U':'A', 'G':'C', 'C':'G'}
gencode = {
'TTT': 'F', 'TTC': 'F', 'TTA': 'L', 'TTG': 'L', 'TCT': 'S',
'TCC': 'S', 'TCA': 'S', 'TCG': 'S', 'TAT': 'Y', 'TAC': 'Y',
'TGT': 'C', 'TGC': 'C', 'TGG': 'W', 'CTT': 'L', 'CTC': 'L',
'CTA': 'L', 'CTG': 'L', 'CCT': 'P', 'CCC': 'P', 'CCA': 'P',
'CCG': 'P', 'CAT': 'H', 'CAC': 'H', 'CAA': 'Q', 'CAG': 'Q',
'CGT': 'R', 'CGC': 'R', 'CGA': 'R', 'CGG': 'R', 'ATT': 'I',
'ATC': 'I', 'ATA': 'I', 'ATG': 'M', 'ACT': 'T', 'ACC': 'T',
'ACA': 'T', 'ACG': 'T', 'AAT': 'N', 'AAC': 'N', 'AAA': 'K',
'AAG': 'K', 'AGT': 'S', 'AGC': 'S', 'AGA': 'R', 'AGG': 'R',
'GTT': 'V', 'GTC': 'V', 'GTA': 'V', 'GTG': 'V', 'GCT': 'A',
'GCC': 'A', 'GCA': 'A', 'GCG': 'A', 'GAT': 'D', 'GAC': 'D',
'GAA': 'E', 'GAG': 'E', 'GGT': 'G', 'GGC': 'G', 'GGA': 'G',
'GGG': 'G', 'TAA': 'STOP', 'TAG': 'STOP', 'TGA': 'STOP'}
Problema: qual a sequência da proteína codificada por
AGCTGGATCCTGAACGATGCATAAGCATAGCCATAGACTAGCATGGGACTAAAGGTCCATTACTGA
Sabemos que o módulo biosequences tem um dicionário chamado gencode.
from biosequences import gencode def translation(seq): cods = [seq[i:i+3] for i in range(0, len(seq), 3)] prot = [] for c in cods: aa = gencode[c] if aa == 'STOP': break prot.append(aa) return ''.join(prot) seq = 'AGCTGGATCCTGAACGATGCATAAGCATAGCCATAGACTAGCATGGGACTAAAGGTCCATTACTGA' print(seq) print(translation(seq))
AGCTGGATCCTGAACGATGCATAAGCATAGCCATAGACTAGCATGGGACTAAAGGTCCATTACTGA SWILNDA
Claro que a função translation() seria uma boa adição ao nosso módulo
biosequences...
O projeto BioPython foi precisamente criado como uma coleção de funções e objetos que suportam a representação e transformação de sequências biológicas.
Hoje é muito mais do que isso, mas a parte central deste pacote de módulos continua a ser a funcionalidade relacionada com sequências biológicas.
Se usarmos a primeira forma do comando import, é possível mudar o nome
do módulo (para uma forma mais abreviada), um alias, da seginte forma:
import biosequences as bs h, s = bs.readFASTA("gre3.txt") print(f'Header:\n{h}\n\nSequence:\n{s}')
Header: >sp|P38715|GRE3_YEAST NADPH-dependent aldose reductase GRE3 OS=Saccharomyces cerevisiae (strain ATCC 204508 / S288c) GN=GRE3 PE=1 SV=1 Sequence: MSSLVTLNNGLKMPLVGLGCWKIDKKVCANQIYEAIKLGYRLFDGACDYGNEKEVGEGIRKAISEGLVSRKDIFVVSKLWNNFHHPDHVKLALKKTLSDMGLDYLDLYYIHFPIAFKYVPFEEKYPPGFYTGADDEKKGHITEAHVPIIDTYRALEECVDEGLIKSIGVSNFQGSLIQDLLRGCRIKPVALQIEHHPYLTQEHLVEFCKLHDIQVVAYSSFGPQSFIEMDLQLAKTTPTLFENDVIKKVSQNHPGSTTSQVLLRWATQRGIAVIPKSSKKERLLGNLEIEKKFTLTEQELKDISALNANIRFNDPWTWLDGKFPTFA